MEM de etiquetado por proximidad (APEX-MS)
Para este enfoque, la proteína de interés se fusiona con una peroxidasa (ácido ascórbico peroxidasa; APEX). Tras el tratamiento con H2O2, la APEX convierte el biotin-fenol suministrado exógenamente en radicales biotin-fenoxilo, lo que da lugar a un etiquetado covalente de la proteína en un radio de 20nm. Posteriormente, las proteínas biotiniladas se enriquecen con Steptavidina. Esta tecnología permite por primera vez tomar «instantáneas» de los entornos locales de las proteínas en un momento dado y capturar las interacciones transitorias de las proteínas. Los enfoques de etiquetado basados en la proximidad han avanzado rápidamente en los últimos años y se ha demostrado que proporcionan resultados complementarios a los métodos tradicionales de purificación basados en la afinidad (AP-MS), por lo que tienen el potencial de abrir vías de investigación totalmente nuevas y proporcionar nuevos conocimientos sobre las funciones de las proteínas.
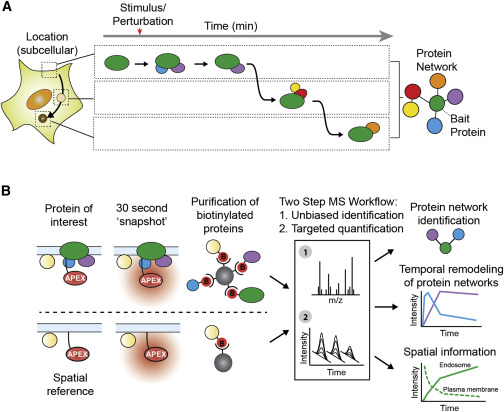
La APEX se ha utilizado para capturar proteomas de orgánulos enteros con alta resolución temporal, pero generalmente se piensa que su amplitud de etiquetado impide la mayor resolución espacial necesaria para interrogar redes de proteínas específicas. Recientemente hemos aportado una solución a este problema combinando la proteómica cuantitativa con un sistema de referencias espaciales. Este novedoso método nos permite resolver los socios de unión conocidos, así como los componentes de la red no identificados previamente, como se ha demostrado recientemente en un estudio de prueba de principio que interroga a las proteínas involucradas por los receptores acoplados a proteínas G a medida que señalan y trafican dinámicamente en respuesta a la activación inducida por el ligando.