Proximity-Labeling MS (APEX-MS)
Pour cette approche, la protéine d’intérêt est fusionnée à une peroxydase (acide ascorbique peroxydase ; APEX). Lors du traitement avec H2O2, l’APEX convertit le biotine-phénol fourni de manière exogène en radicaux biotine-phénoxyle, ce qui entraîne un marquage covalent de la protéine dans un rayon de 20nm. Ensuite, les protéines biotinylées sont enrichies à l’aide de Steptavidine. Cette technologie permet pour la première fois de prendre des « instantanés » de l’environnement local des protéines à un moment donné et de capturer les interactions protéiques transitoires. Les approches de marquage de proximité ont progressé rapidement au cours des dernières années et il a été démontré qu’elles fournissent des résultats complémentaires aux méthodes traditionnelles de purification par affinité (AP-MS), ayant ainsi le potentiel d’ouvrir des voies de recherche entièrement nouvelles et de fournir de nouveaux aperçus des fonctions des protéines.
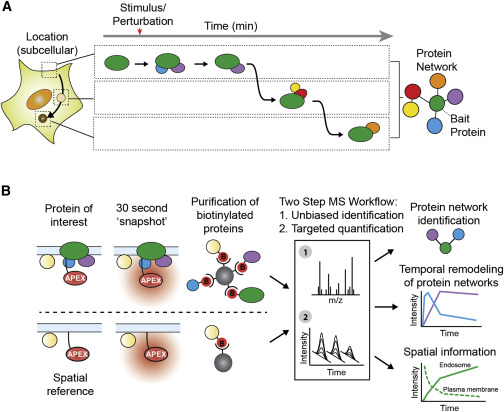
APEX a été utilisé pour capturer des protéomes d’organites entiers avec une haute résolution temporelle, mais sa largeur de marquage est généralement considérée comme excluant la résolution spatiale plus élevée nécessaire pour interroger des réseaux de protéines spécifiques. Nous avons récemment apporté une solution à ce problème en combinant la protéomique quantitative avec un système de références spatiales. Cette nouvelle méthode nous permet de résoudre les partenaires de liaison connus, ainsi que les composants de réseau précédemment non identifiés, comme cela a été récemment démontré dans une étude de preuve de principe interrogeant les protéines engagées par les récepteurs couplés aux protéines G alors qu’ils signalent et circulent dynamiquement en réponse à l’activation induite par le ligand.