Proximity-Labeling MS (APEX-MS)
Per questo approccio la proteina di interesse è fusa a una perossidasi (acido ascorbico perossidasi; APEX). Dopo il trattamento con H2O2, APEX converte la biotina-fenolo fornita esogenamente in radicali biotina-fenossile, che risulta in un’etichettatura covalente della proteina in un raggio di 20nm. Successivamente, le proteine biotinilate sono arricchite utilizzando Steptavidina. Questa tecnologia permette per la prima volta di prendere “istantanee” di ambienti proteici locali in un dato momento e di catturare interazioni proteiche transitorie. Gli approcci di etichettatura basati sulla prossimità sono avanzati rapidamente negli ultimi anni e hanno dimostrato di fornire risultati complementari ai tradizionali metodi di purificazione basati sull’affinità (AP-MS), avendo così il potenziale per aprire strade completamente nuove di ricerca e fornire nuove intuizioni sulle funzioni delle proteine.
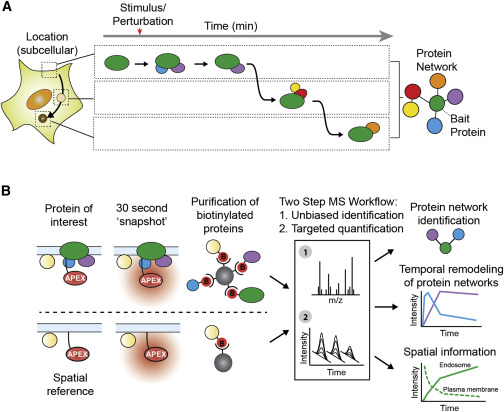
APEX è stato utilizzato per catturare interi proteomi organello con alta risoluzione temporale, ma la sua ampiezza di etichettatura è generalmente pensato per precludere la risoluzione spaziale superiore necessaria per interrogare reti specifiche proteine. Abbiamo recentemente fornito una soluzione a questo problema combinando la proteomica quantitativa con un sistema di riferimenti spaziali. Questo nuovo metodo ci permette di risolvere i partner di legame noti, così come i componenti di rete precedentemente non identificati, come recentemente dimostrato in una prova di principio studio interrogando le proteine impegnate da G-protein-coupled recettori come essi dinamicamente segnale e il traffico in risposta all’attivazione indotta ligando.