Proximity-Labeling MS (APEX-MS)
W tym podejściu interesujące nas białko jest sprzężone z peroksydazą (peroksydaza kwasu askorbinowego; APEX). Po potraktowaniu H2O2, APEX przekształca egzogennie dostarczoną biotynę-fenol do rodników biotynowych-fenoksylowych, co powoduje kowalencyjne znakowanie białka w promieniu 20nm. Następnie, biotynylowane białka są wzbogacane przy użyciu Steptavidin. Technologia ta pozwala po raz pierwszy na wykonanie „migawek” lokalnych środowisk białkowych w danym czasie i uchwycenie przejściowych interakcji białkowych. W ciągu ostatnich kilku lat nastąpił szybki rozwój metod znakowania opartych na bliskości i wykazano, że dostarczają one wyników komplementarnych do tradycyjnych metod oczyszczania opartych na powinowactwie (AP-MS), dzięki czemu mogą otworzyć zupełnie nowe drogi badań i zapewnić nowy wgląd w funkcje białek.
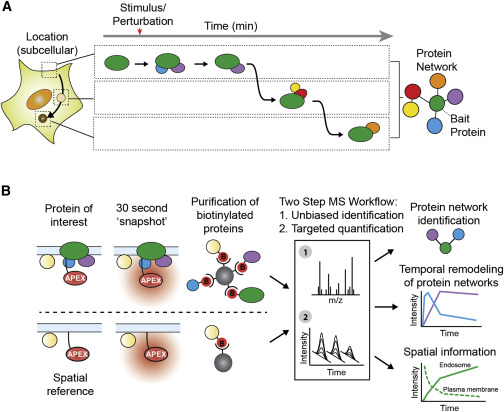
APEX był używany do wychwytywania całych proteomów organelli z wysoką rozdzielczością czasową, ale jego szeroki zakres znakowania jest ogólnie uważany za wykluczający wyższą rozdzielczość przestrzenną niezbędną do badania specyficznych sieci białek. Niedawno zaproponowaliśmy rozwiązanie tego problemu poprzez połączenie proteomiki ilościowej z systemem odniesień przestrzennych. Ta nowatorska metoda pozwala nam określić znanych partnerów wiążących, jak również wcześniej niezidentyfikowane komponenty sieci, jak pokazano ostatnio w badaniu proof of principle, w którym badano białka zaangażowane przez receptory sprzężone z białkami G, gdy dynamicznie sygnalizują i przemieszczają się w odpowiedzi na aktywację wywołaną przez ligand.
.