Etiquetagem de Proximidade da EM (APEX-MS)
Para esta abordagem a proteína de interesse é fundida com uma peroxidase (ácido ascórbico peroxidase; APEX). No tratamento com H2O2, o APEX converte os radicais biotina-fenol fornecidos exogenamente em biotina-fenoxil, o que resulta numa rotulagem covalente da proteína num raio de 20 nm. Posteriormente, as proteínas biotiniladas são enriquecidas com Steptavidin. Esta tecnologia permite, pela primeira vez, tirar “instantâneos” de ambientes proteicos locais num determinado momento e capturar interacções proteicas transitórias. As abordagens de rotulagem baseadas na proximidade avançaram rapidamente nos últimos anos e demonstraram fornecer resultados complementares aos métodos tradicionais de purificação baseados na afinidade (AP-MS), tendo assim o potencial de abrir caminhos inteiramente novos de pesquisa e fornecer novos conhecimentos sobre as funções proteicas.
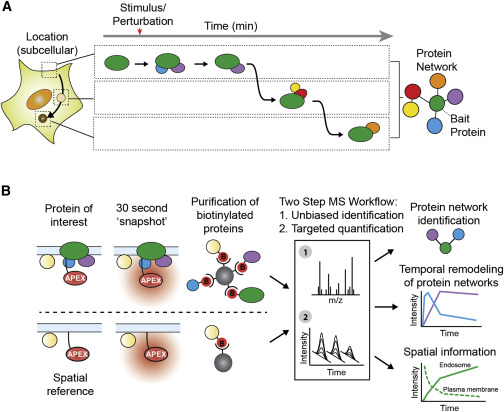
APEX tem sido usado para capturar proteomas organela inteiros com alta resolução temporal, mas pensa-se geralmente que a sua amplitude de rotulagem impede a maior resolução espacial necessária para interrogar redes proteicas específicas. Recentemente fornecemos uma solução para este problema, combinando proteômica quantitativa com um sistema de referências espaciais. Este novo método permite-nos resolver parceiros de ligação conhecidos, bem como componentes de rede previamente não identificados, como mostrado recentemente num estudo de prova de princípio interrogando proteínas envolvidas por receptores acoplados à proteína G à medida que estes sinalizam dinamicamente e tráfego em resposta à activação induzida pela ligação.